研究者根据自身的研究需要,针对感兴趣的基因功能确立基因列表,或者基于已有的其他组学的结果,确定感兴趣的基因集。注意,如果后续是需要在小鼠等非人类细胞系中进行筛选的话,需要相应的小鼠基因列表。我们以人的基因列表为例。
CRISPick是博德研究所(Broad Institute)发布的sgRNA在线设计工具,包括基因敲除(CRISPRko)、激活(CRISPRa)和沉默(CRISPRi)三种设计模块,物种也囊括了人、小鼠以及大鼠等模式物种,其中人和小鼠还提供了不同参考基因组的不同版本。研究者可以根据自身的实验目的及实验需要进行相应的sgRNA设计。该工具根据大规模的实验室筛选结果结合深度学习,对sgRNA的切割效率及脱靶情况进行估计,从而给出预测结果。CRISPick是目前最为常用的sgRNA设计工具。
研究者根据自身的研究需要,针对感兴趣的基因功能确立基因列表,或者基于已有的其他组学的结果,确定感兴趣的基因集。注意,如果后续是需要在小鼠等非人类细胞系中进行筛选的话,需要相应的小鼠基因列表。我们以人的基因列表为例。
CRISPick提供了三种CRISPR的设计模式,以敲除(CRISPRko)为例,目前主要有2种系统用于筛选,即:单质粒系统和双质粒系统。如果您选择双质粒系统,那么在筛选前,需要将表达Cas9的质粒转入待筛选的细胞系。这里以单质粒系统lentiCRISPR v2为例,介绍以下的设计流程。
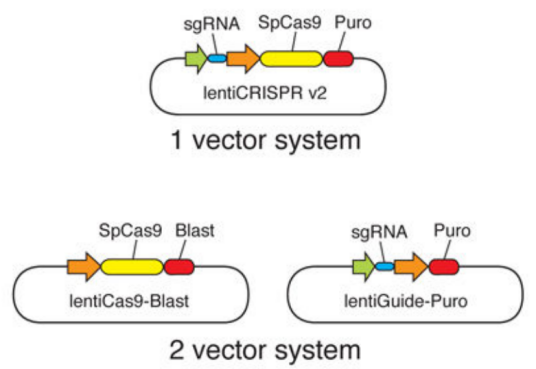
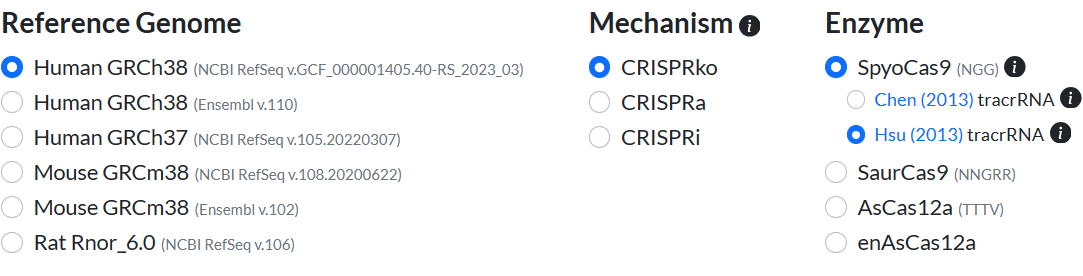
进入CRISPick的网站:CRISPick (broadinstitute.org),参考基因组这里选择Human GRCh38,筛选模式选择CRISPRko。由于lentiCRISPR v2的tracrRNA是以GTTTT开头的,酶这里选择SpyoCas9中的Hsu(2013)tracrRNA版本。如果您的载体中tracrRNA是以GTTTV(V代表任意碱基)开头的,则可以选择Chen(2013)tracrRNA版本。
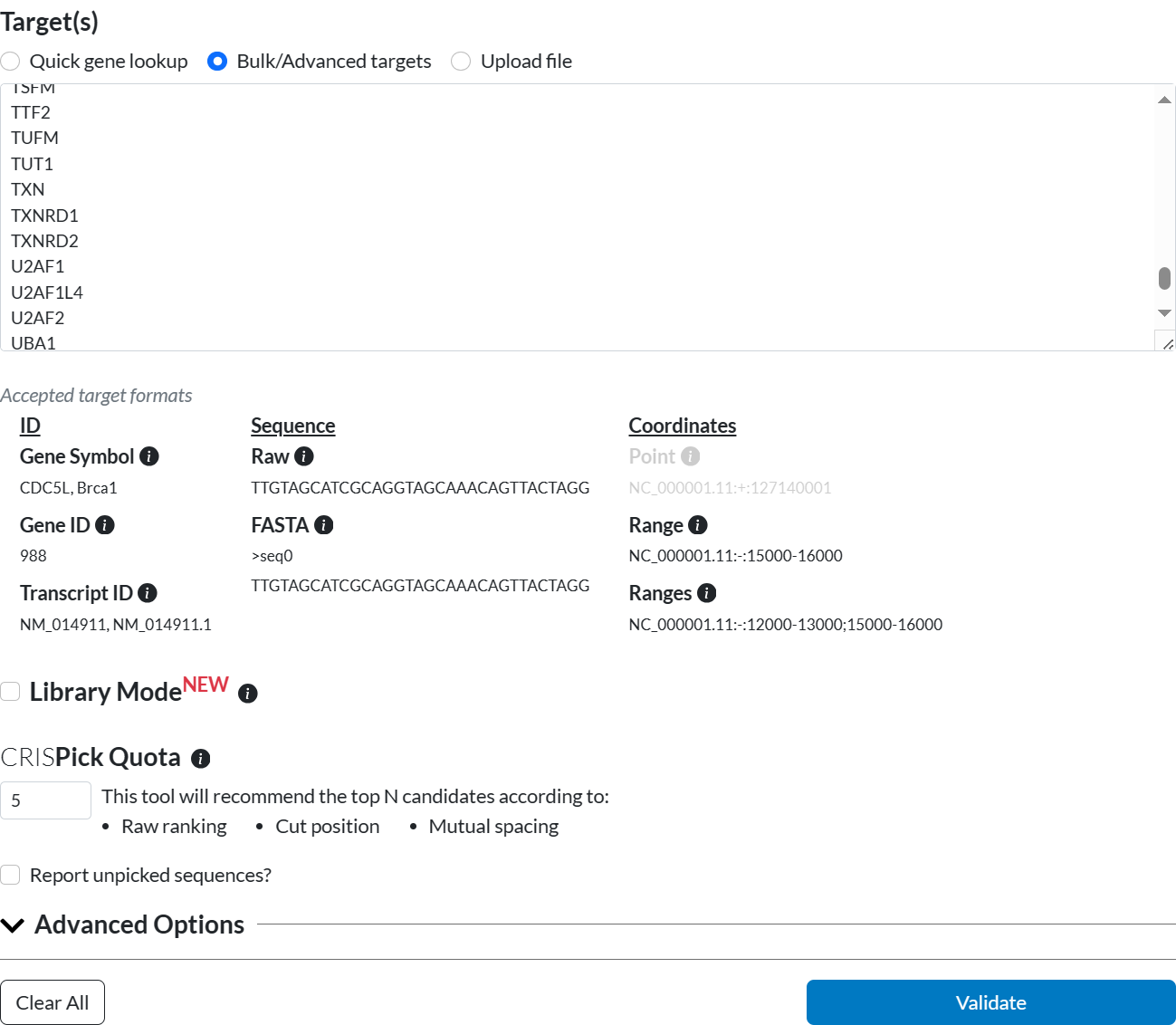
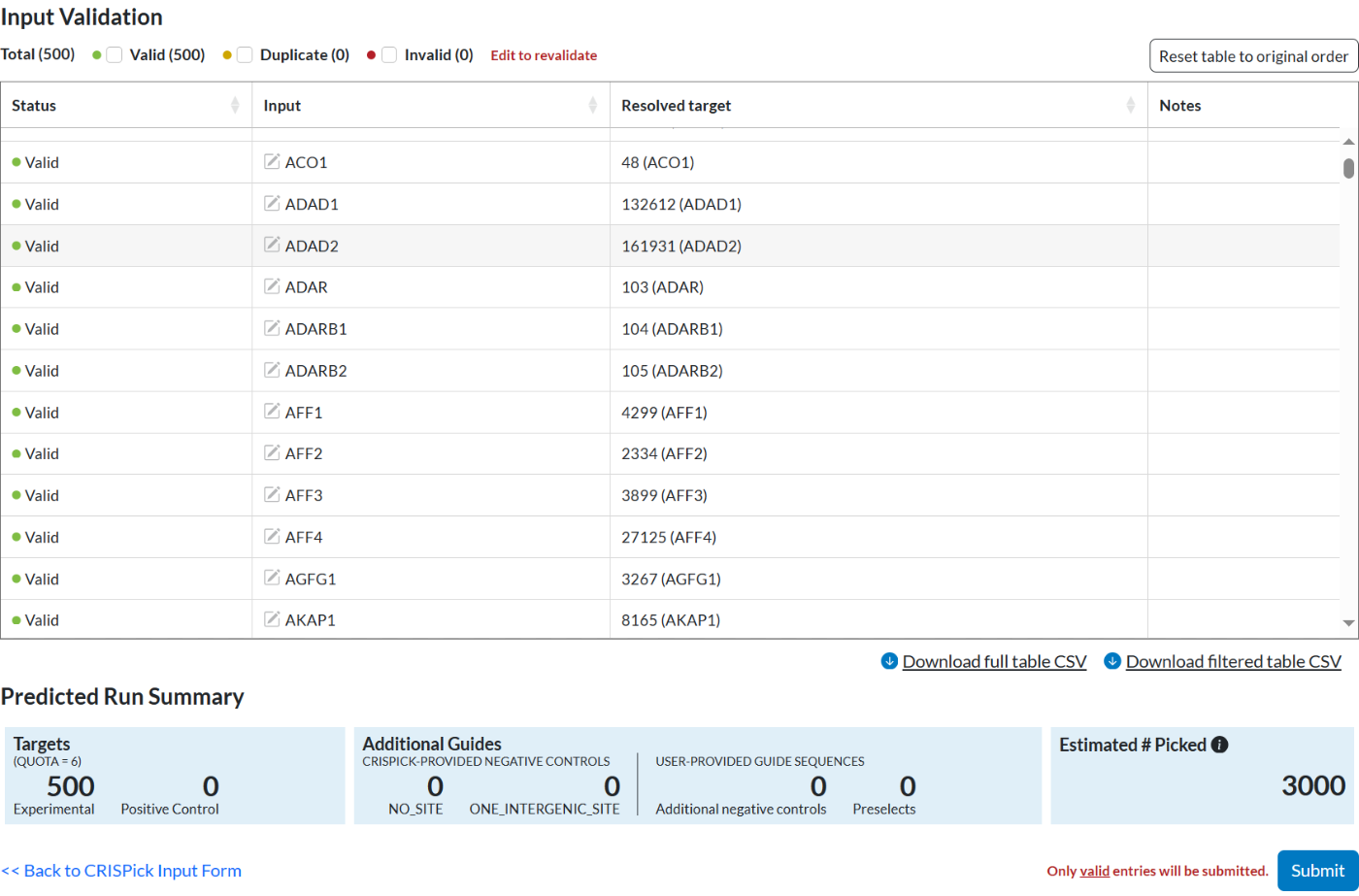
CRISPick的Bulk/Advanced targets模式可以批量设计sgRNA,但是每次最多设计500个基因的sgRNA。单个基因也提供快速查看模式Quick gene lookup。由于前期准备的基因名称可能与CRISPick中使用的名称有差异而导致设计失败,这里需要预提交以确认正确的基因名称。这里将整个基因列表的基因全部输入。然后点击最下方的Validate,进入下一步确认。
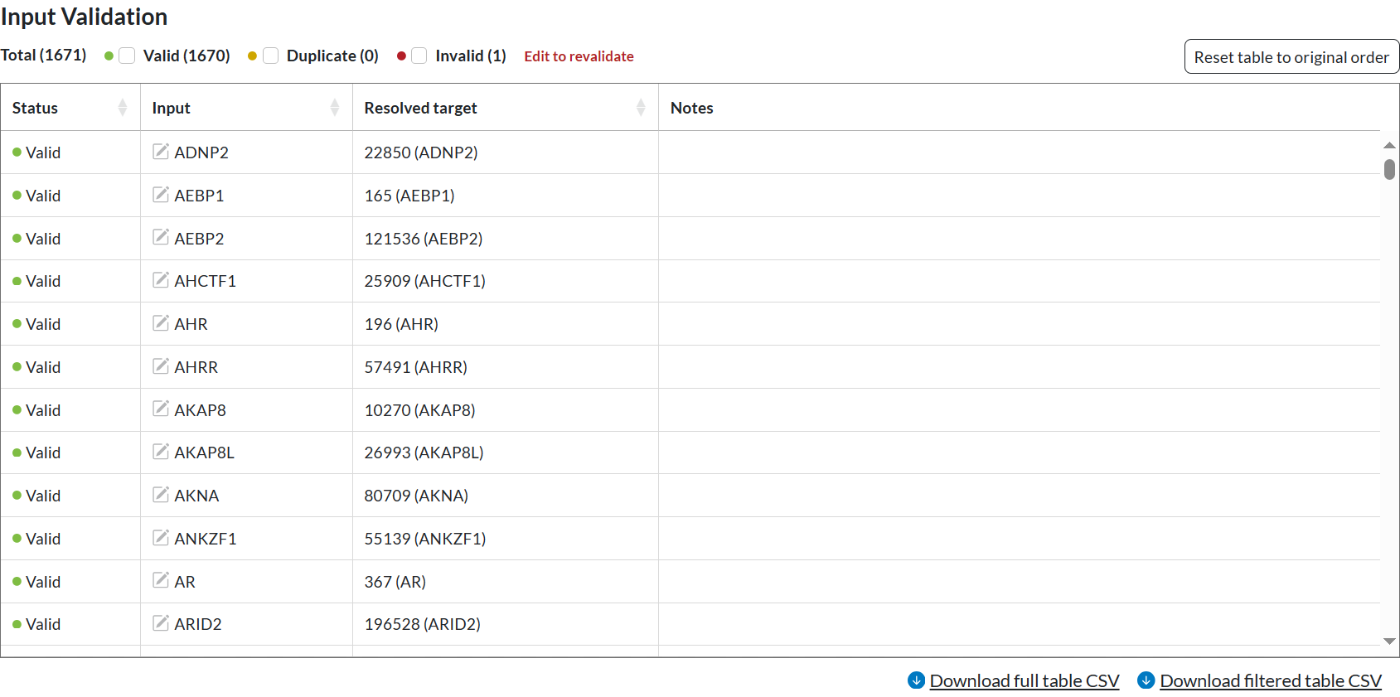
如果有基因重复的情况或者找不到的情况,可以在Duplicate和Invalid中查阅。尤其是综合不同来源的基因时,有些基因可能同时对应不同的名称,很容易造成重复。还有的基因名称跟参考基因组版本不一致导致找不到。这时可以通过Genecard或者HGNC等数据库合适基因的名称,利用Quick gene lookup模式检查是否正确。实在查不到的基因可以利用原始DNA序列设计sgRNA,或者极少数基因可以放弃亦可。
将完善好的基因列表再次分批提交,每次最多提交500个基因。根据需要输入每个基因需要设计多少条sgRNA。这里选择为每个基因设计6条sgRNA,然后确认。

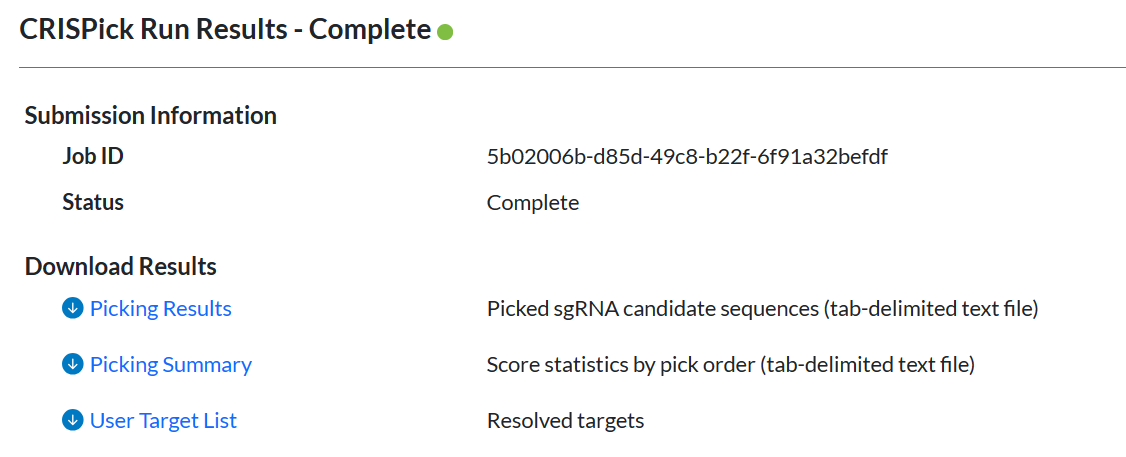
等待任务完成后点击Picking Results下载结果。综合几次提交的结果,检查sgRNA序列是否有重复。如果有重复的sgRNA需要单独重新设计新的sgRNA,最终保证文库中所有sgRNA没有重复。同时,需要为文库加入一定数量的negative control sgRNA。必要时也可以添加positive control sgRNA。
以Gibson Assembly构建法为例,在合成引物池之前,需要为sgRNA两端添加同源臂各20bp以及扩增引物池的引物序列。此同源臂根据研究者所选的载体来设计。最终将设计好的引物池序列进行合成用于下游的构建实验。